Coronavírus no mundo.
Um vírus que foi relatado pela primeira vez na cidade chinesa de Wuhan, agora se espalhou para mais de uma dúzia de países em todo o mundo, provocando uma crise econômica e de saúde sem precedentes. A Organização Mundial da Saúde (OMS) declarou o surto do coronavírus uma emergência de saúde pública de interesse mundial.
Vamos dá uma breve olhada na crise atual e depois nos aprofundaremos no “Novel Corona Virus 2019 Dataset” do Kaggle.
O que é um coronavírus?
Segundo a OMS, os coronavírus (CoV) são uma grande família de vírus que causam doenças que variam do resfriado comum a doenças mais graves, como a Síndrome Respiratória do Oriente Médio (MERS-CoV) e a Síndrome Respiratória Aguda Grave (SARS-CoV). Um novo coronavírus (nCoV) é uma nova cepa que não foi previamente identificada em humanos. O vírus identificado como a causa do surto recente está sendo chamado de coronavírus 2019-nCoV ou Wuhan.
A crise, a partir de hoje
De acordo com o último relatório do New York Times, “o número de infecções confirmadas subiu para 37.198 e o número de mortos na China aumentou para 811, superando o número de mortos pela epidemia de SARS.”
Dezesseis cidades na China, com uma população combinada de mais de 50 milhões de pessoas, estão confinadas. As companhias aéreas de todo o mundo cancelaram voos de ida e volta dá China.
Alguns países estão evacuando seus cidadãos em vôos especiais e os colocando em quarentena rigorosa (Brasil por exemplo). Para piorar, as bolsas de valores caíram na China e os mercados em todo o mundo estão sentindo os efeitos. Alguns analistas prevêem que o surto representa uma ameaça para a economia global, e que tem o potencial de desencadear conseqüências geopolíticas de longo alcance.
Uma introdução ao conjunto de dados
O Conjunto de dados do “Novel Corona Virus 2019”, publicado no Kaggle, foi coletado pela John Hopkins University. A equipe coletou os dados de várias fontes, como a OMS, CDC local e meios de comunicação. Eles também criaram um painel em tempo real para monitorar a propagação do vírus.
Importando e Carregando os Dados
|
|
Compreendendo o conjunto de dados
Vamos primeiro obter um entendimento básico do conjunto de dados e executar operações de limpeza de dados, se necessário.
|
|
## Sno Date Province.State Country Last.Update Confirmed
## 1 1 01/22/2020 12:00:00 Anhui China 01/22/2020 12:00:00 1
## 2 2 01/22/2020 12:00:00 Beijing China 01/22/2020 12:00:00 14
## 3 3 01/22/2020 12:00:00 Chongqing China 01/22/2020 12:00:00 6
## 4 4 01/22/2020 12:00:00 Fujian China 01/22/2020 12:00:00 1
## 5 5 01/22/2020 12:00:00 Gansu China 01/22/2020 12:00:00 0
## 6 6 01/22/2020 12:00:00 Guangdong China 01/22/2020 12:00:00 26
## Deaths Recovered
## 1 0 0
## 2 0 0
## 3 0 0
## 4 0 0
## 5 0 0
## 6 0 0
Os nomes das colunas são auto-explicativos. A primeira coluna ‘Sno’ se parece com um número de linha e não agrega valor à análise. A quinta coluna ‘Última atualização’ mostra o mesmo valor que a coluna ‘Data’, exceto em alguns casos em que os números foram atualizados posteriormente. Então, irei remover essas duas colunas antes de continuar.
|
|
## Date Province.State Country Confirmed Deaths Recovered
## 1 01/22/2020 12:00:00 Anhui China 1 0 0
## 2 01/22/2020 12:00:00 Beijing China 14 0 0
## 3 01/22/2020 12:00:00 Chongqing China 6 0 0
## 4 01/22/2020 12:00:00 Fujian China 1 0 0
## 5 01/22/2020 12:00:00 Gansu China 0 0 0
## 6 01/22/2020 12:00:00 Guangdong China 26 0 0
Se fizer uma análise mais aprofundada, a base de dados mostra que faltam nomes de províncias para países como Reino Unido, França e Índia. Nesse caso, não podemos assumir ou preencher valores ausentes de nenhuma observação.
|
|
## Date Province.State Country
## 02/14/2020 22:00:00: 75 : 418 Mainland China:739
## 02/15/2020 22:00:00: 75 Anhui : 25 US :166
## 02/13/2020 21:15:00: 74 Beijing : 25 Australia : 76
## 02/11/2020 20:44:00: 73 Chongqing: 25 Canada : 53
## 02/12/2020 22:00:00: 73 Fujian : 25 China : 34
## 02/07/2020 20:24:00: 72 Gansu : 25 Japan : 25
## (Other) :1127 (Other) :1026 (Other) :476
## Confirmed Deaths Recovered
## Min. : 0 Min. : 0.000 Min. : 0.00
## 1st Qu.: 2 1st Qu.: 0.000 1st Qu.: 0.00
## Median : 12 Median : 0.000 Median : 0.00
## Mean : 406 Mean : 9.121 Mean : 33.66
## 3rd Qu.: 101 3rd Qu.: 0.000 3rd Qu.: 5.00
## Max. :56249 Max. :1596.000 Max. :5623.00
##
Fazendo uma rápida análise descritiva, usando a função summary()
A função summary() retorna as estatísticas gerais das colunas da base de
dados. Uma conclusão imediata da saída é que os dados foram relatados
cumulativamente, ou seja, o número de casos relatados em um determinado
dia inclui os casos relatados anteriormente. O valor ‘máximo’ de mortes
é 1596, o que é consistente com os relatos da mídia há alguns dias
(quando esses dados foram publicados
15/02/2020).
|
|
## [1] China US Japan
## [4] Thailand South Korea Mainland China
## [7] Hong Kong Macau Taiwan
## [10] Singapore Philippines Malaysia
## [13] Vietnam Australia Mexico
## [16] Brazil France Nepal
## [19] Canada Cambodia Sri Lanka
## [22] Ivory Coast Germany Finland
## [25] United Arab Emirates India Italy
## [28] Sweden Russia Spain
## [31] UK Belgium Others
## [34] Egypt
## 34 Levels: Australia Belgium Brazil Cambodia Canada China Egypt ... Vietnam
Os dados mostram que o vírus se espalhou para 33 países da Ásia, Europa e América. Para facilitar essa análise, podemos mesclar dados para ‘China’ e ‘Mainland China’. (Others não é um país e sim a contagem dos valores em brancos).
|
|
## [1] China US Japan
## [4] Thailand South Korea Hong Kong
## [7] Macau Taiwan Singapore
## [10] Philippines Malaysia Vietnam
## [13] Australia Mexico Brazil
## [16] France Nepal Canada
## [19] Cambodia Sri Lanka Ivory Coast
## [22] Germany Finland United Arab Emirates
## [25] India Italy Sweden
## [28] Russia Spain UK
## [31] Belgium Others Egypt
## 34 Levels: Australia Belgium Brazil Cambodia Canada China Egypt ... Vietnam
Antes de avançar, vamos verificar o formato das datas na coluna ‘Data’.
|
|
## [1] 01/22/2020 12:00:00 01/22/2020 12:00:00 01/22/2020 12:00:00
## [4] 01/22/2020 12:00:00 01/22/2020 12:00:00 01/22/2020 12:00:00
## 25 Levels: 01/22/2020 12:00:00 01/23/2020 12:00:00 ... 02/15/2020 22:00:00
Parece que os dados foram atualizados em horários diferentes a cada dia. Podemos extrair as datas do registro de data e hora e usá-las para análises adicionais. Isso nos ajudará a manter as datas uniformes.
|
|
## [1] "2020-01-22" "2020-01-22" "2020-01-22" "2020-01-22" "2020-01-22"
## [6] "2020-01-22"
Agora vamos ter uma noção do impacto do surto em cada país.
|
|
## # A tibble: 33 x 4
## Country Confirmed Deaths Recovered
## <fct> <dbl> <dbl> <dbl>
## 1 Australia 5 0 4
## 2 Belgium 1 0 0
## 3 Brazil 0 0 0
## 4 Cambodia 1 0 1
## 5 Canada 4 0 1
## 6 China 56249 1596 5623
## 7 Egypt 1 0 0
## 8 Finland 1 0 1
## 9 France 12 1 4
## 10 Germany 16 0 1
## # … with 23 more rows
-
Alto número de casos na China
-
Alto número de mortes na China
-
China e França foram os únicos países com mortes confirmadas
-
Não houve casos confirmados no Brasil
Plotando os dados
Para visualização dos dados, usaremos três poderosos pacotes do R: ggplot2, dplyr e tidyr.
1 - Número de casos confirmados ao longo do tempo
|
|
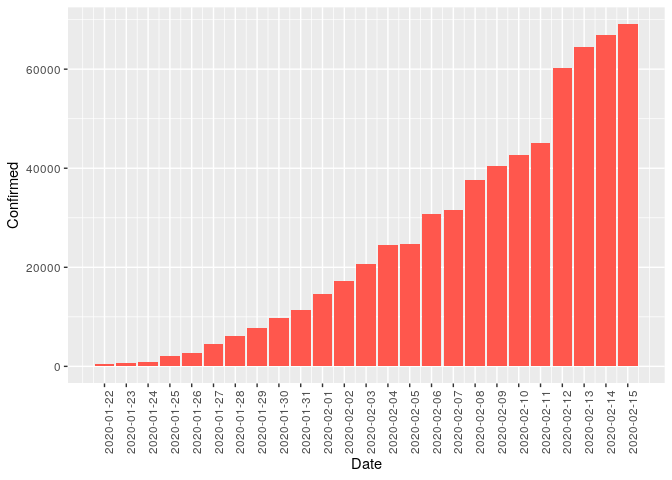
Pelo gráfico acima, conseguimos ver mais de 60000 casos confirmados.
1.2 - Número de mortes confirmados ao longo do tempo
|
|
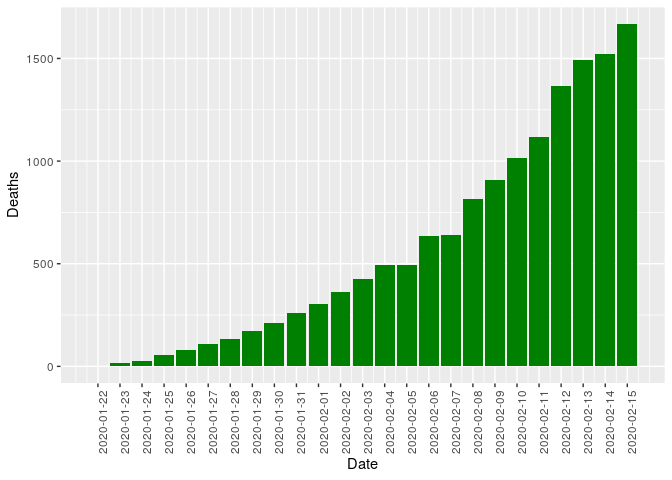
Pelo gráfico acima, conseguimos ver mais de 1500 mortes.
1.3 - Número de casos recuperado ao longo do tempo
|
|
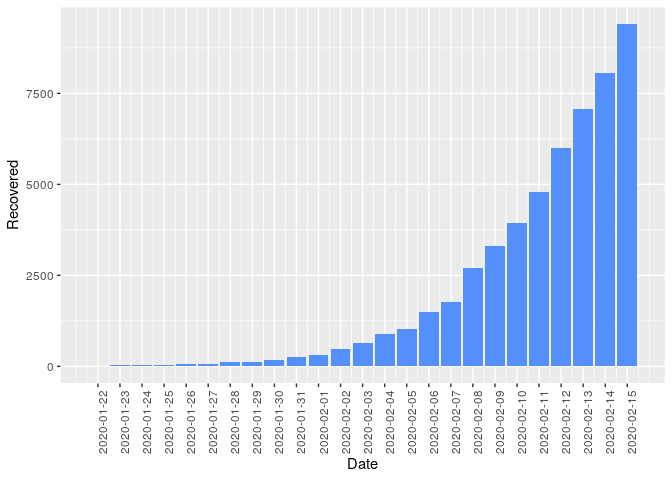
Pelo gráfico acima, conseguimos ver mais de 7500 casos de pessoas recuperadas.
1.4 - Todos os casos ao longo do tempo
|
|
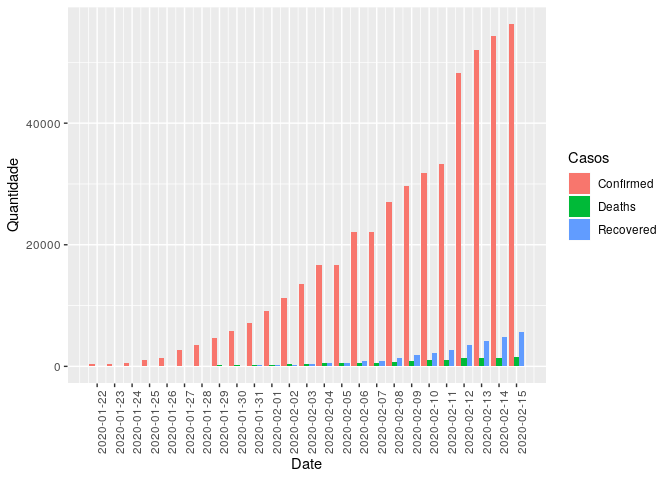
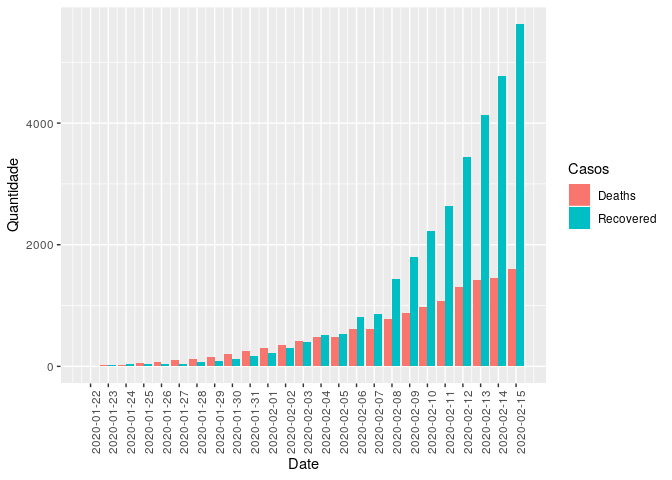
- Mortes e Recuperados
Diante todo o caos, podemos retirar uma noticia boa. Os casos de pessoas recuperadas já superam e muito o número de mortos.
|
|
- Um olhar mais atento às 10 províncias mais afetadas da China
|
|
## # A tibble: 34 x 4
## Province.State Confirmed Deaths Recovered
## <fct> <dbl> <dbl> <dbl>
## 1 Hubei 56249 1596 5623
## 2 Guangdong 1294 2 410
## 3 Henan 1212 13 391
## 4 Zhejiang 1162 0 428
## 5 Hunan 1001 2 425
## 6 Anhui 950 6 221
## 7 Jiangxi 913 1 210
## 8 Jiangsu 604 0 186
## 9 Chongqing 544 5 184
## 10 Shandong 532 2 156
## # … with 24 more rows
Observações
-
A província chinesa de Hubei é o epicentro do surto. Tem significativamente mais casos relatados do que todas as outras províncias juntas. Existem algumas províncias onde não houve mortes e todos os pacientes afetados se recuperaram.
-
O número de casos relatados diariamente aumentou quase 500% desde 22 de janeiro. Isso mostra que o vírus é altamente contagioso e está se espalhando rapidamente.
-
Durante a primeira semana, a taxa de mortes foi superior à das recuperações. Desde 31 de janeiro, a taxa de recuperação disparou e está mostrando uma tendência positiva. Houve 255 recuperações no dia 4 de fevereiro, em comparação com 66 mortes. A taxa de recuperação continuará a aumentar à medida que mais pessoas conhecerem os sintomas e forem rápidas na procura de medicamentos.
-
Países geograficamente próximos à China, como Tailândia, Japão e Cingapura, relataram mais casos do que outros países asiáticos e europeus. A Alemanha é uma exceção e tem o maior número de casos na Europa.
Conclusão
A análise mostra a taxa alarmante na qual o coronavírus está se espalhando. Pelo menos 1590 pessoas já morreram durante a atual epidemia, excedendo as 774 mortes relatadas durante o surto de SARS há sete anos.